陈巍学基因A04-A06
- 简单序列比对
- 基础Boyer-Moore算法
陈巍学基因A04-A06
A04 简单序列比对
在这个实践中,会写一个(碱基)完全匹配的算法,并用它把人工做出来的read比对到基因组上。
首先先下载一个PhiX生物体(PhiX是一种噬菌体)的基因组开始:
wget --no-check https://d28rh4a8wq0iu5.cloudfront.net/ads1/data/phix.fa
#encoding:utf-8
def readGenome(filename):
genome = ''
with open(filename,'r') as f:
for line in f:
if not line[0] == '>':
genome += line.rstrip()
return genome
genome = readGenome('phix.fa')
#p作为read,t作为基因组
def naive(p,t):
occurrences = []
for i in range(len(t) - len(p) + 1):
match = True
for j in range(len(p)):
if not t[i+j] == p[j]:
match = False
break
if match:
occurrences.append(i)
return occurrences
t = 'AGCTTAGAtAGC'
p = 'AG'
print(naive(p,t))
# 从genome中随机产生子序列
import random
def generateReads(genome,numReads,readLen):
'''Generate reads from random positions in the given genome.'''
reads = []
for _ in range(numReads):
start = random.randint(0,len(genome)-readLen) - 1
reads.append(genome[start : start+readLen])
return reads
reads = generateReads(genome,100,100)
numMatched = 0
for r in reads:
matches = naive(r,genome)
if len(matches)>0:
numMatched +=1
print("%d / %d reads matched exactly!" % (numMatched,len(reads)))
A05 简单序列比对法的效率
实际进行比较的次数与可能比较次数的最小值接近
A06 基础Boyer-Moore算法
跟上面定义的naive算法接近,但可以跳过很多不必要的比较,是算是精确比较算法中的标杆了,应用十分广泛

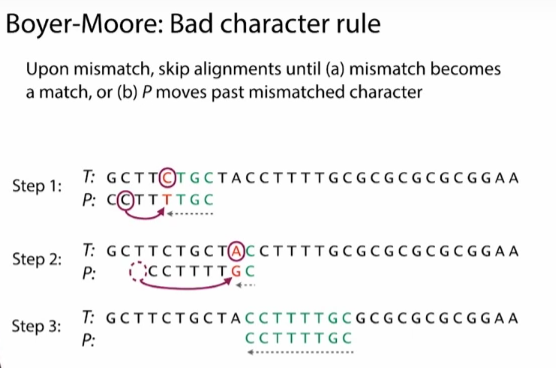
由于u没有在
P中出现过,所以,我们直接跳过U!而Boyer-Moore算法将这个规则应用到最大限度。
Try alignments in left-to-right order,and try character comparisons in right-to-left order

Bad character rule:当我们遇到错配(mismatch)时,我们移动P,直到错配变成匹配 用坏字规则有2步,这也是用Boyer-Moore算法的规则之一,
好后缀规则:让
t等于内部循环中,匹配的子字符串好字规则是针对我们能够匹配上的字符的,我们要移动P,移动的结果,是不会把匹配的,变成错配的
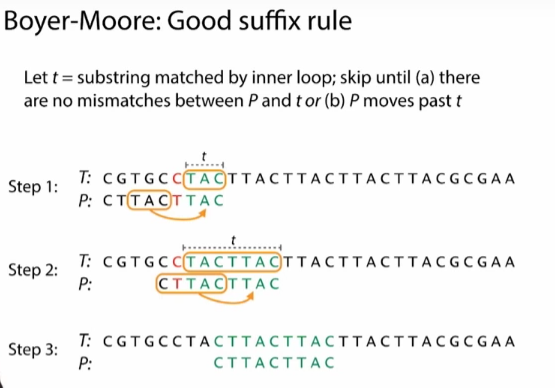
坏字符规则把不匹配的转到匹配的,好后缀规则试图把匹配上,保持匹配的状态,让它不要变成错配的。