陈巍学基因A01-A03
噬菌体基因组简单探究
陈巍学基因A01-A03
A01:查看基因组的信息
下载一个噬菌体的基因组
wget --no-check https://d28rh4a8wq0iu5.cloudfront.net/ads1/data/lambda_virus.fa
#encoding:utf-8
def readGenome(filename):
genome = ''
with open(filename,'r') as f:
for line in f:
if not line[0] == '>':
genome += line.rstrip()#rstrip是去除跟在字符串后面的空白
return genome
genome = readGenome('lambda_virus.fa')
print("the length of 噬菌体genome is:\n")
print(len(genome))
#统计碱基个数
counts = {'A':0,'C':0,'T':0,'G':0}
for base in genome:
counts[base] += 1
print(counts)
####使用模块计算####
#Counter会遍历字符串中的每个字符,并且对每个字符出现次数进行计数
import collections
t=collections.Counter(genome)
print(t)
A02: 查看fastq 的信息
对于fastq文件,可以看到以四行为单位, 第一行以@符号开头,这是一个tag,是个哪个序列的名字。 第二行是DNA碱基的字符串 第三行是+号 第四行是read的质量序列
下载一个fastq: wget --no-check https://d28rh4a8wq0iu5.cloudfront.net/ads1/data/SRR835775_1.first1000.fastq
#encoding:utf-8
#将测序的结果及测序的质量分别放到俩数组里
def readFastq(filename):
sequences = []
qualities = []
with open(filename) as fh:
while True:
fh.readline()
seq = fh.readline().rstrip()
fh.readline()
qual = fh.readline().rstrip()
if len(seq) == 0:
break
sequences.append(seq)
qualities.append(qual)
return sequences,qualities
seqs,quals = readFastq('SRR835775_1.first1000.fastq')
#将phred 33 编码转成数值的质量分数
def phred33ToQ(qual):
return ord(qual) - 33
def createHist(qualities):
hist = [0] * 50
for qual in qualities:
for phred in qual:
q = phred33ToQ(phred)
hist[q] += 1
return hist
h = createHist(quals)
# 使用matplotlib(一个Python上的图形化工具)来做(可视化)
import matplotlib as mpl
mpl.use('Agg')
import matplotlib.pyplot as plt
plt.bar(range(len(h)),h)
plt.savefig('1.png')
将phred 33 编码的值转成质量分数,也就是测序质量,将ASCII字符转变成质量分数 ord函数取一个字符,并把该字符的ASCII值编程数值 print(phred33ToQ('#')) #得到2,说明测序质量很低。可信度比较低。 错误的可能性大概有30~32%,且'#'多出现在read的末尾,说明督导read的末尾时,确信度会下降
最低为2,最高为44
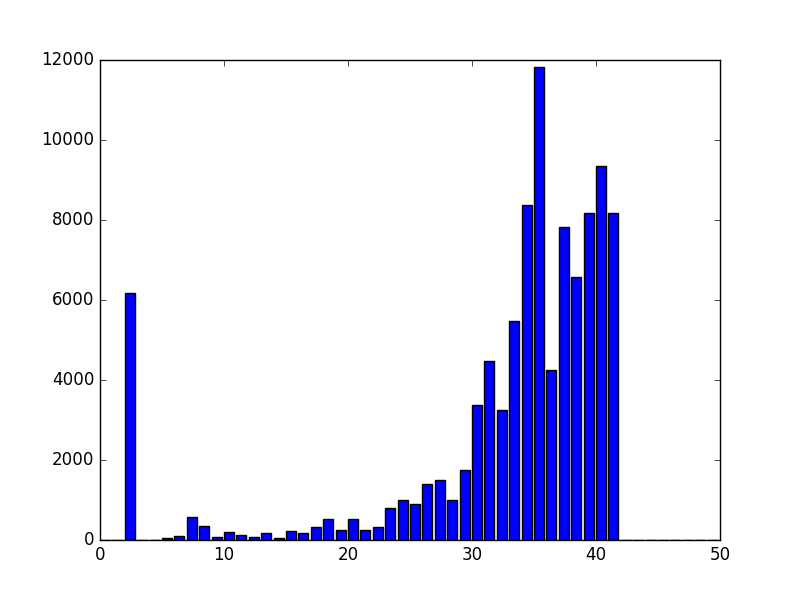
从图形结果来看,分值为2的很多,说明这一部分是质量比较差的,可能原因是这一部分的碱基发出的荧光是四种颜色的混合,比较难确认。
matplotlib小代码
import matplotlib as mpl
mpl.use('Agg')
import matplotlib.pyplot as plt
plt.bar(range(len(h)),h)
plt.savefig('1.png')
必须关闭上个图形设备才能保存新画的图形:
fig = plt.figure(0) # 新图 0
plt.savefig() # 保存
plt. close(0) # 关闭图 0
或者
fig = plt.figure() # 新图 0
plt.savefig() # 保存
plt.close('all') # 关闭图 0
A03:按碱基位置分析GC含量
不同的物种会有不同的GC含量,把整个read的GC做成图形,来看测序中是否有什么strange的事情发生。
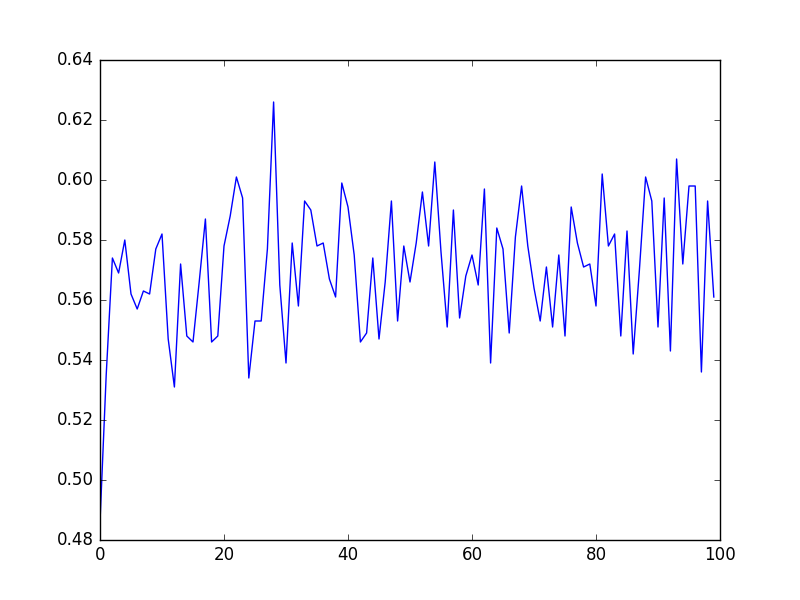
图的GC含量大概60~80%
import collections
count = collections.Counter()
for seq in seqs:
count.update(seq)
print(count)
Counter({'G': 28742, 'C': 28272, 'T': 21836, 'A': 21132, 'N': 18})
有一些N:当碱基判读程序对一个碱基的判读很不确定时,它就会读成"N"