陈巍学基因A07-A09
- 应用Boyer-Moore的规则
- 把参考基因组做成索引
陈巍学基因A07-A09
A07:应用Boyer-Moore 二规则
我们需要联合使用坏字规则和好后缀规则。只要碰到错配,我们就要同时使用这2个规则。两个规则都会告诉我们需要跳过的次数,我们只要选择最大的进行跳过就行了。
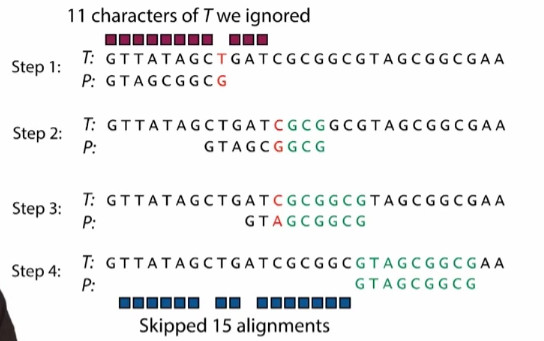
实际在实施Boyer-Moore算法时,要预先建一个查询表,这样每当我们要用任何一个规则时,就到这个表里去查找可以跳多少步。做这个表格只需要模式P而不需要文本T。好后缀,坏字都需要创建表格。
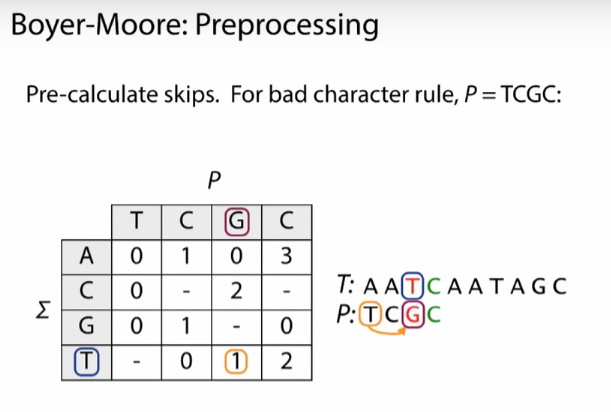
A08:
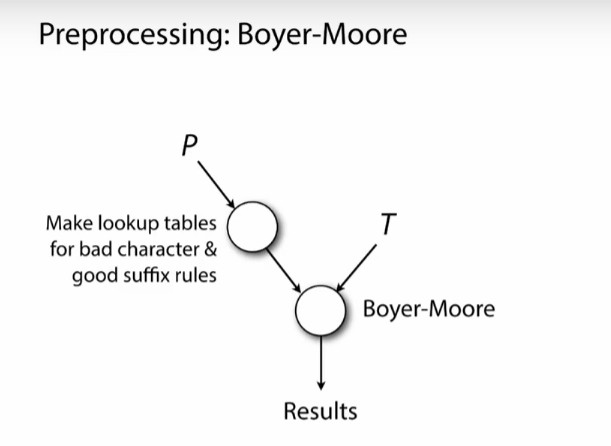
预处理可能会花费较多的时间,但均摊到每次比对,消耗就比较少了。
同样的,我们可以预处理T,解决重复比对的问题。
把文本T进行预处理版本的算法,叫做离线算法(offline algorithm),而不处理文本的算法,被称为online。在线算法还是离线算法,与我们是否预处理P无关。区别在于,是否预处理文本T
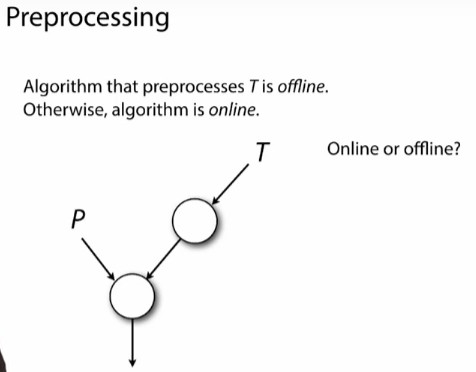
- naive算法是在线算法,还是离线算法?在线。naive算法完全不做任何预处理。
- Boyer-Moore算法是在线还是离线?在线。只对P进行了预处理。
- Web search engine?离线算法。
- Read alignment 基因序列比对问题?离线算法
A09:把参考基因组做成索引
把参考基因组看成是一本书,我们可以给书加上索引。当想查找某个单词,直接看索引就行了。是根据字母进行排序的。
另一个例子:到杂货店买牛奶,找到奶制品,再找牛奶。是按照分类进行排序的。
- 做一个长度为5的子字符串的偏移值串的索引。在一张表中,放下所有
K-mer的偏移量。
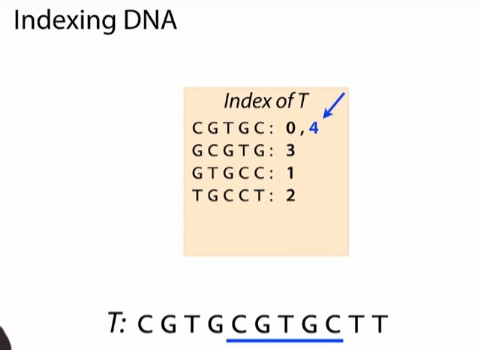
关于第二个3,是按照字典顺序,将第三个索引插到了0和1之间。
关于第一行的4,是添加一个新的值。
做好索引:

我们用K-mer:substring of length K用K-mer来指代长度为K的子字符串。对于长度为5的子字符串,我们说5-mer。
- 如何查询这个索引?query the index
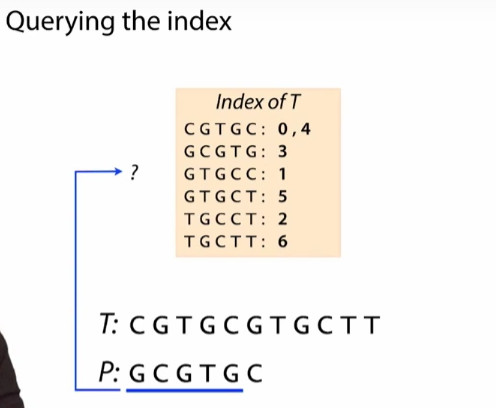
我们找到了3
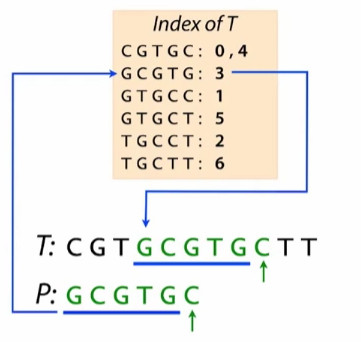
下一步验证第六个碱基是否与T相同。相同,我们可以得出结论,P在T偏移3个碱基的位置。
- 同样的,我们也可以从右边开始进行查询:
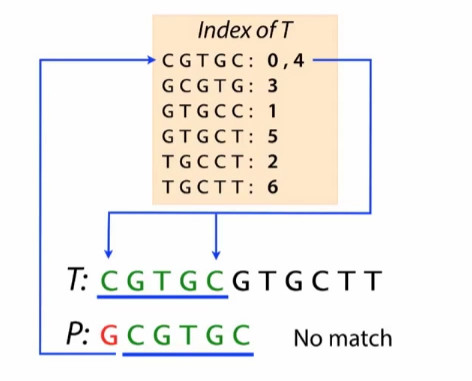
当查询到偏移为0的时候,由于0左边没有碱基,不能与P的第一位match,所以查询失败。
在查询偏移值为4:match!
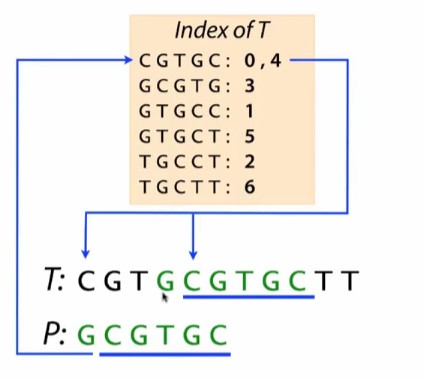
说明在使用该模式时,用哪个
5-mer并不重要,它们中的任何一个都会引导出正确的P和T匹配的偏移值。
- 一个修改的例子:修改后P不再match T。
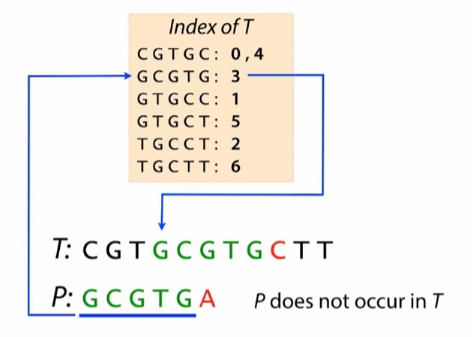
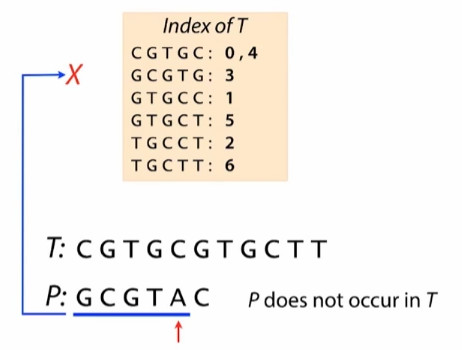
定义index 相匹配为hit,hit说明有可能match ,也有可能不match。