modele_name
title: 无root安装UCSC genome browser author: woobamboo authorURL: authorFBID:
如何在本地实现一个定制化的UCSC genome browser?
1. 安装必要的软件
需要安装Apache 及 MySQL (版本5.x), 其中Apache可以更换成Nginx,MySQL后面直接同步UCSC的数据库到本地,因此不要更改。
MySQL有编译好的版本,Apache编译好的版本已经比较老了。两者的安装方法很简单,请看前面的文章。
另外,由于UCSC部分代码是C语言实现的,请确保你的glibc版本!!!至少大于等于``GLIBC_2.17,你应该能通过下面的命令看到GLIBC_2.17`:
检查glibc版本
$strings /lib64/libc.so.6 |grep GLIBC
版本太低会报错:/lib64/libc.so.6: version 'GLIBC_2.14' not found , /lib64/libc.so.6: version 'GLIBC_2.17' not found
2. 请确保一下事情已经做好:
glibc大于等于2.17Apache已安装并正常运行MySQL(v5.x)已安装并正常运行,再次强调版本是5.x
3. 几个目录的说明:
# Apache 的安装目录
drwxr-xr-x. 2 dongkaige rnomics 39 Dec 11 10:10 logs
drwxr-xr-x. 9 dongkaige rnomics 4.0K Dec 10 20:43 htdocs
drwxr-xr-x. 4 dongkaige rnomics 145 Dec 8 20:31 conf
drwxr-xr-x. 2 dongkaige rnomics 4.0K Dec 8 02:28 modules
drwxr-xr-x. 2 dongkaige rnomics 74 Dec 7 16:50 cgi-bin
drwxr-xr-x. 2 dongkaige rnomics 4.0K Dec 2 23:02 bin
......
- 几个重要目录
htdocs这个目录就是你网站的默认根目录。安装好Apache之后,访问的第一个网页就在这里放着!confApache的配置目录,前期要经常修改配置文件。cgi-bin存放的是可执行程序,由网页调用。这是安装好Apache之后的默认可执行程序存放路径,我们后面会修改。比如你写了个helloWorld.py,正确的配置将使它显示执行结果而不是在网页显示你的代码。logs这个文件很有用!显示Apache执行日志以及错误日志;bin存放Apache主程序,后面可能经常用到./apachectl restartmodules:存放MySQL默认编译好的模块,后面我们自己编译的模块也需要放到这里面。
# MySQL 的安装目录
-rw-r-----. 1 dongkaige rnomics 145K Dec 11 12:13 error.log
drwxr-x---. 11 dongkaige rnomics 4.0K Dec 10 14:45 data
-rw-r--r--. 1 dongkaige rnomics 552 Dec 9 14:19 my.cnf
drwxr-xr-x. 2 dongkaige rnomics 4.0K Dec 7 16:02 bin
...
- 几个重要目录
my.cnfMySQL配置文件,一般配置好了就不再修改了。dataMySQL 数据库文件,里面一个文件夹就是一个数据库!binMySQL 主程序,常用语法就那么几个,看前面博客。
4. 下载静态文件,让UCSC主页显示出来!
前面说过,Apache下的htdocs 目录存放的是网页信息,我们在其中下载静态网页:
cd htdocs && mkdir genomebrowser
rsync -avzP rsync://hgdownload.cse.ucsc.edu/htdocs/ genomebrowser/
# 然后,在htdocs目录下(一些网页引用资源的相对位置在其本身的上层目录):
ln -s genomebrowser/js
ln -s genomebrowser/style
ln -s genomebrowser/images
# 下载完之后,浏览器打开http://your.url:port/genomebrowser 将能显示浏览器主页!
# 在genomebrowser文件夹 新建一个cgi-bin,并下载网页后台调用的程序:
mkdir genomebrowser/cgi-bin
rsync -avzP rsync://hgdownload.cse.ucsc.edu/cgi-bin/ genomebrowser/cgi-bin/
参考这个图,理解下面的内容: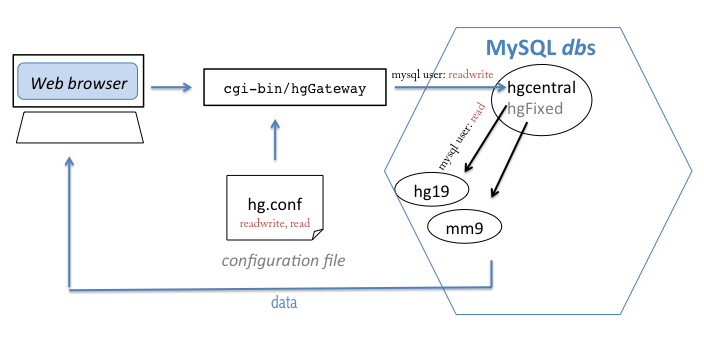
让Apache找到可执行程序
现在静态文件已经能显示出来了,但导航栏是用cgi-bin程序实现的,现在并不能看到。我们需要配置Apache,让它知道到如何找到这些可执行程序,这里需要配置httpd.conf (Apache 的配置文件),添加下面内容:
ScriptAlias /genomebrowser/cgi-bin /your/path/to/apache_2.4.37/htdocs/genomebrowser/cgi-bin
<Directory "/your/path/to/apache_2.4.37/htdocs/genomebrowser/cgi-bin">
AllowOverride None
#Options +ExecCGI -MultiViews +SymLinksIfOwnerMatch
Options +ExecCGI
Order allow,deny
Allow from all
AddHandler cgi-script .cgi .pl .py
Require all granted
</Directory>
大意就是添加一个可执行程序的位置,并将几种后缀认为是可执行程序(
.cgi,.pi,.py没必要添加.py,我是为了测试用),并允许这个文件夹下内容访问远程资源。
另外在httpd.conf中添加下面内容使得网页正确显示:
XBitHack on
<Directory /your/pach/to/genomebrowser>
AllowOverride AuthConfig
Options +Includes
</Directory>
现在,Apache已经知道如何找到UCSC的可执行程序了,还需要进一步配置,让可执行程序找到MySQL的数据:
让可执行程序访问MySQL
hg.conf 这个文件至关重要,应用程序通过它访问MySQL。可以下载个样本:http://genome-test.cse.ucsc.edu/~kent/src/unzipped/product/ex.hg.conf 看看。
在配置hg.conf之前,先进行MySQL的配置(按前面博客),然后建立三个用户,使之拥有不同权限:
ucsc_read: 只读权限的用户:ucsc_readwrite: 对一个数据库具有读写权限的用户,因为cgi-bin程序需要更新hgcentral来维护用户的cookie设置ucsc_admin: 一个MySQL用户,具有所有browser和genome数据库的权限:
# 创建三个用户,并赋予不同权限
GRANT ALL PRIVILEGES ON *.* TO 'ucsc_admin'@'%';
GRANT SELECT ON *.* TO 'ucsc_read'@'%';
GRANT ALL PRIVILEGES ON hgcentral TO 'ucsc_readwrite'@'%';
flush PRIVILEGES;
hg.conf配置如下:
db.host=10.10.123.123 # 填你自己的。这里写localhost似乎不行,因为这样写会导致port(端口) 的设置无效!
db.socket=/picb/rnomics2/RBP_database/dbweb/Softwares/mysql_5/mysql.sock # MySQL运行时的sock文件!
db.port=7090 # 访问MySQL的端口
db.user=ucsc_read # 填你自己的MySQL用户名
db.password=PASSWORD
db.trackDb=trackDb # trackDb 是MySQL中 hgcentral 数据库的一个表,这个数据库后面再创建,这里主要是让UCSC能访问MySQL!
db.grp=grp
defaultGenome=Human
central.host=10.10.123.123
central.socket=/picb/rnomics2/RBP_database/dbweb/Softwares/mysql_5/mysql.sock # MySQL运行时的sock文件!
central.port=7090 # 访问MySQL的端口
central.user=ucsc_readwrite
central.password=PASSWORD
central.db=hgcentral # 我们后面会创建这个数据库:hgcentral
central.domain=
重要说明:
因为我们本地非root安装了MySQL,运行MySQL的端口和
sock文件是自己指定的,所以hg.conf一定要指定端口和sock文件才能正确连接!及时查看Apache的log文件,查看MySQL的连接状态。
central.socket与central.port虽然别的参考里都没有提,但是就是这里没有设置引起的错误困扰了我很久。参考db.socket与db.port于是新增了central的两个设置。
然后重启Apache,试试看能否显示导航栏!要是不能,需要检查前面的步骤,进行排错。(因为导航栏是由可执行程序进行显示的,如果上面没设置好,是看不到导航栏的。)
~/.hg.conf文件
UCSC 提供了一些程序来方便的管理MySQL数据库,这些程序通过访问~/.hg.conf来连接MySQL。这些程序可由源代码编译,或直接从linux.x86_64下载得到。位于cgi-bin不使用该配置文件。
.hg.conf配置如下:
db.host=10.10.123.123
db.socket=/path/to/your/mysql.sock
db.port=your_mysql_port
db.user=ucsc_admin
db.password=ucsc_admin_passwd
.hg.conf的权限应该是-rw———,否则报错。
现在服务器应该已经能访问MySQL了,但是MySQL里还没有任何数据,我们下载一些进行测试:
5. 配置MySQL服务器
UCSC需要的数据在两个位置进行存储
- 大部分数据,如
注释存储在MySQL数据库中; - 有些数据,例如
参考基因组是2bit 格式,直接存放在硬盘里,这个位置被编码进MySQL表,默认为/gbdb,且没有直接配置它位置的选项。后面我们可以通过更新MySQL表来修改,大部分是在hgcentral.dbDB中。
MySQL数据库中的hgcentral: 用来存储其他数据库的信息。请参考这个图理解hgcentral: 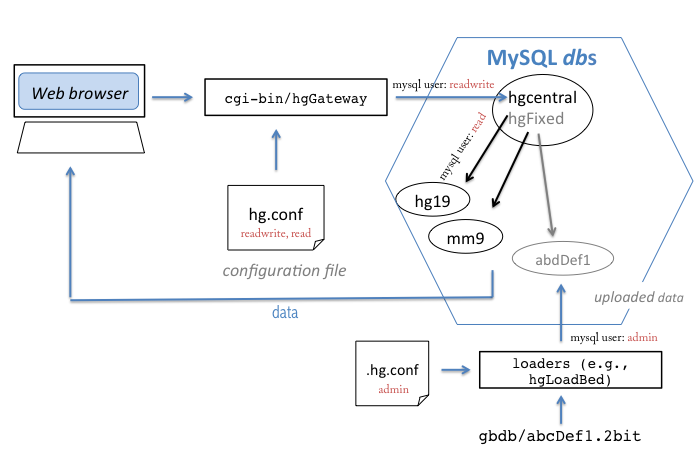
# 建立关键数据库 hgcentral
wget http://hgdownload.cse.ucsc.edu/admin/hgcentral.sql
# 自己修改hgcentral.sql 将 /gbdb/ 替换成你自己的 /your/path/to/gbdb/ 路径
sed 's/\/gbdb\//\/your\/path\/to\/gbdb\//g' hgcentral.sql > hgcentral_new.sql
mysql –u用户名 –p密码 -e "create database hgcentral"
mysql –u用户名 –p密码 hgcentral < hgcentral_new.sql
# 给你自己的MySQL用户操作这个数据库的权限
这里通过修改
hgcentral.sql,来解决它的gbdb设置都是在根目录的问题。
MySQL数据库的作用
- hgcentral
- hg19
数据库hgcentral包含其他数据库的信息,hgcentral.dbDb.nibPath (hgcentral的dbDb表的nibPath字段),它相当于网关火地址簿;另一个表hgcentral.genomeClade控制基因组浏览器上一些信息。
hgcentral看起来像是这样:
+---------------------+
| Tables_in_hgcentral |
+---------------------+
| blatServers |
| clade |
| dbDb |
| dbDbArch |
| defaultDb |
| gbMembers |
| genomeClade |
| hubPublic |
| hubStatus |
| liftOverChain |
| namedSessionDb |
| sessionDb |
| targetDb |
| userDb |
+---------------------+
14 rows in set (0.01 sec)
MySQL 中每个参考基因组都有自己的数据库,在每个基因组数据库中,都有一些必要的特定名称的表格。annotation表是上传track创建的,后面讨论。对于你的基因组,数据库看起来应该像这样:
+------------------------+
| Tables_in_abcDef1 |
+------------------------+
| Custom_Annotation_1 |
| Custom_Annotation_2 |
| chromInfo |
| gap |
| gc5Base |
| gold |
| grp |
| hgFindSpec |
| history |
| trackDb |
+------------------------+
trackDb包含Custom_Annotation_1, Custom_Annotation_2和其他你自己制作的track的信息。
上面简要说明了MySQL中的数据库,下面还需要/gbdb/后面再说。
一个例子
为了节省时间出来进行排错,这里选取一个小数据集(dm3),首先下载一些MySQL数据表:
cd /path/to/mysql/data
mkdir dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/chromInfo.MYD /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/chromInfo.MYI /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/chromInfo.frm /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/cytoBandIdeo.MYD /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/cytoBandIdeo.MYI /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/cytoBandIdeo.frm /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/grp.MYD /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/grp.MYI /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/grp.frm /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/hgFindSpec.MYD /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/hgFindSpec.MYI /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/hgFindSpec.frm /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/trackDb.MYD /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/trackDb.MYI /home/mysql/dm3
rsync -avzP rsync://hgdownload.cse.ucsc.edu/mysql/dm3/trackDb.frm /home/mysql/dm3
# dm3文件夹是个数据库,每三个文件是一个表,具体后缀的含义,自己查。
重启MySQL,你将能看到dm3 这个数据库已经存在在MySQL里了(我用Navicat软件可视化操作的)。
本来按照教程,我们应该准备gbdb文件了(是不能或者不适合保存在MySQL数据库的文件,如二进制的基因组、bigwig文件等。):
# 下载gbdb数据: (大概6 G) 在这个位置创建的 /gbdb 无法直接在浏览器里显示,需要修改MySQL里的hgcentral数据库的相关表,其中存储的有比如基因组位置;默认是/gbdb/dm3/??.2bit 这样的格式,是在根目录的。因为是非Root,所以还是老实修改表吧。我上面创建 hgcentral 数据库时已经修改了表。
mkdir -p genomebrowser/gbdb/dm3
rsync -avzP --delete --max-delete=20 rsync://hgdownload.cse.ucsc.edu/gbdb/dm3/ /home/genomebrowser/gbdb/dm3/
建议下载之前看看有多大,命令在后面。
但是因为网速问题,这就不下了。但为了看看前面做的怎么样,还是看下效果:在你的浏览器里打开 http://your/url:port/genomebrows/cgi-bin/hgGateway?db=dm3
和 http://your/url:port/genomebrows/cgi-bin/hgTracks?db=dm3
这个URL 透漏了很多信息:就是执行cgi-bin下的hgGateway 文件,进行查询,看数据库里有没有dm3这个东西!
最后开始添加Custom Track !这个步骤,对于理解整个流程至关重要,如果前面一头雾水,这里要实际操作一下!
gbdb文件夹 这个文件夹我花了很长时间才搞懂(主要是前面各种错误用了了很多时间)。这个文件夹是为了存储一些二进制文件,不方便存储在MySQL的文件。应用程序如何找到这些二进制文件呢?其实,这些文件的位置是作为数据存放在hgcentral数据库的表里的。对于默认的基因组,比如hg19,它们的位置(也就是MySQL表里存放的hg19基因组的位置)是/gbdb/hg19/hg19.2bit,是从根目录开始的,对于你自己的custom track来说,因为上传track时需要指定路径,则不需要修改MySQL表格,就能正确显示。这个直接把数据位置写进到MySQL的做法,被下面一个引用称为猴子才会做的事情。这篇文章还被UCSC官方推荐为参考之一。。
6. trash 文件夹
这里主要用来存放临时数据、临时track的,对我来说没什么用,就没配置。同样需要在hg.conf中配置其位置。详细看后面的参考链接。
7. MySQL的SSL配置
UCSC教程里有,我没有做
8. 添加Custom Track
其实非常简单:最简单只需要三步,但前面还是要做很多事情:
首先下载一些程序:这些程序能读取~/.hg.conf 来访问我们的MySQL数据库,从而方便将track数据写到MySQL表中。其中大部分程序用不上,但是还是都下了吧:
git clone git://genome-source.cse.ucsc.edu/kent.git # 这个其实是cgi-bin的源代码,一会会用到里面的sql语句
rsync -azvP rsync://hgdownload.soe.ucsc.edu/genome/admin/exe/linux.x86_64/ ./
修改了配置文件,才能正确访问MySQL:(这个配置文件一定要在~/也就是主目录下,没找到如何自定义它的位置的参数。)
~/.hg.conf 如下配置:
db.host=10.10.123.123
db.socket=/mysqlPath/mysql_5/mysql.sock
db.port=yourPort
db.user=your_mysql_user
db.password=your_mysql_password
central.host=10.10.123.123
central.socket=/mysqlPath/mysql_5/mysql.sock
central.port=your_port
central.user=your_mysql_user
central.password=your_mysql_password
central.db=hgcentral
这样,UCSC提前写好的程序就能通过~/.hg.conf 访问我们的数据库了。
本来按照教程,
linux.x86_64里有个hgsql,能让我们hgsql -e "show databases;" mysql这样优雅的,不用输密码的访问MySQL数据的,但是我的有问题,只好把教程里的类似步骤都换成MySQL语法,手动去做了。
然后,比如有个bigwig文件,放在gbd/hg38/bw/a.bw这里,我们想把它上传上去,需要修改/gbdb/hg38下的track.ra文件,
track.ra
track bw2
shortLabel bw2_short
longLabel bw2_long
visibility dense
group map
autoScale on
priority 20
color 62, 62, 62
type bigwig
linux.x86_64/hgTrackDb . hg38 trackDb kent/src/hg/lib/trackDb.sql /picb/rnomics2/RBP_database/dbweb/Softwares/apache_2.4.37/htdocs/genomebrowser/gbdb/hg38
linux.x86_64/hgBbiDbLink hg38 bw1 /picb/rnomics2/RBP_database/dbweb/Softwares/apache_2.4.37/htdocs/genomebrowser/gbdb/hg38/bw/test/ENCFF100HQU.bw
linux.x86_64/hgFindSpec . hg38 hgFindSpec kent/src/hg/lib/hgFindSpec.sql /picb/rnomics2/RBP_database/dbweb/Softwares/apache_2.4.37/htdocs/genomebrowser/gbdb/hg38
第一行意思是,从gbdb/hg38文件的track.ra读取track信息,并插入到hg38数据库的trackDB表格里。第二行是把track.ra 里的bw2这个track的实际路径添加进表格,第三行大概是刷新一下。
其他类型的数据上传,如bed,如2bit,以后再写。
rsync查看大小rsync -hna --stats rsync://hgdownload.soe.ucsc.edu/gbdb/ | egrep "Number of files:|total size is"
MySQL配置过程中,可能要加载很多的模块,后面补充。
参考
- https://genome.ucsc.edu/goldenpath/help/mirrorManual.html
- http://oliverelliott.org/article/bioinformatics/tut_genomebrowser/
- http://blog.sciencenet.cn/blog-723745-569746.html
- http://emb-bioinfo.fsaa.ulaval.ca/bioinfo/html/admin/mirror.html
- file:///Users/kaigedong/Desktop/UCSC%20Genome%20Browser%E5%AE%89%E8%A3%85.pdf
- Installation of a UCSC Genome Browser on a local machine ("mirror")
- Genome Browser source code